摘要:化合物的极性表面积(PSA)是分子表面极性部分的面积值的总和。极性部分通常在诸如氧或氮的杂元素周围,因此可以认为这些部分结构是加在一起的。PSA是描述分子的极性和脂溶性的描述符,例如辛醇/水分配比(logP)。在PSA和膜渗透性的实验数据之间发现了很好的相关性。例如太大PSA(140埃2具有上述化合物)已被经验已知为具有低的膜渗透性。分子构象从定义中可以看出,计算PSA需要分子的三维结构
化合物的极性表面积(PSA)是分子表面极性部分的面积值的总和。极性部分通常在诸如氧或氮的杂元素周围,因此可以认为这些部分结构是加在一起的。
PSA是描述分子的极性和脂溶性的描述符,例如辛醇/水分配比(logP)。在PSA和膜渗透性的实验数据之间发现了很好的相关性。例如太大PSA(140埃2具有上述化合物)已被经验已知为具有低的膜渗透性。
分子构象
从定义中可以看出,计算PSA需要分子的三维结构。由于分子不一定具有一个构象,因此柔性分子可能需要找到几种构象的PSA。。
分子表面计算
必须计算出某个三维结构/构象的分子表面,才能计算出PSA。分子表面是范德华表面,并且缺乏均匀性。
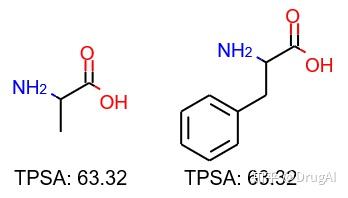
拓扑极性表面积(TPSA)
TPSA中,仅从分子中原子的键合模式(拓扑)估算PSA,而无需考虑分子的三维结构,这是拓扑极性表面积的名称的起源。
TPSA的功能
为了克服PSA计算中的上述问题,TPSA着重于结合模式,并通过将PSA中每个部分的贡献相加来计算PSA。
- 仅考虑耦合模式,可显着降低计算成本
- 以氧,氮,磷,硫为杂元素的统一计算方法
- 通过使用大型药物样化合物数据库进行参数化来提高准确性
考虑到单一构象,计算出世界药品索引中所列化合物的PSA。另外,选择了43个包含氧,氮,磷和硫的部分结构,并且通过最小二乘法对每个部分结构的重量进行参数化以用于计算的PSA。
这样,将某个索引视为来自子结构的贡献之和的方法是一种在化学信息学中经常出现的概念。通过将每个原子分解成亚结构,也可以很容易地看到每个原子的贡献。
导入库
from rdkit import rdBase, Chem
from rdkit.Chem import Draw, Descriptors
from rdkit.Chem.Draw import rdMolDraw2D
from IPython.display import SVG载入数据
suppl=Chem.SDMolSupplier('mol_tpsa.sdf')
mols=[x for x in suppl if x is not None]计算TPSA
declist=Descriptors.descList
calc={}
for (i,j) in declist:
calc[i]=j
tpsa=[calc['TPSA'](i) for i in mols]可视化
view=rdMolDraw2D.MolDraw2DSVG(340,200,170,200)
option=view.drawOptions()
option.legendFontSize=20
option.padding=0.1
view.DrawMolecules(mols, legends=['TPSA: '+str(x) for x in tpsa])
view.FinishDrawing()
svg=view.GetDrawingText()
SVG(svg.replace('svg:', ''))
with open('rdkit_tpsa.svg', 'w') as f:
f.write(svg)